WGCNA分析-生物信息学机器学习研究方法
1.概念
解释:WGCNA分析,中文全称即是加权基因共表达网络分析。该分析方法旨在是寻找协同表达的基因模块,并且去找基因网络和关注的表型之间的联系,以及网络中的核心基因。简单说即用于描述不同样本中基因相关性的模式
实例:例如在之前寻找生姜和胃癌的治疗靶点的时候,利用WGCNA去寻找胃癌差异性表达基因和生姜核心成分的基因寻找出二者的相关性强的的基因模块(一组表达相关性较为强的基因组)
2.原理和方法
2.1首先获得基因相关性表达矩阵
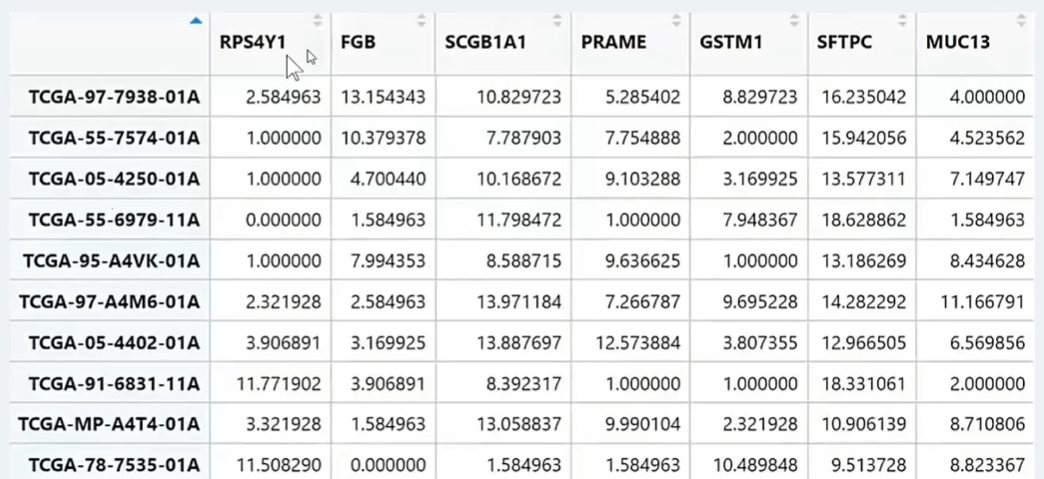
其中第一列为样本名称,第一行是基因名称,将该所有样本的基因表达情况进行相关性的计算,获得相关性表达矩阵表
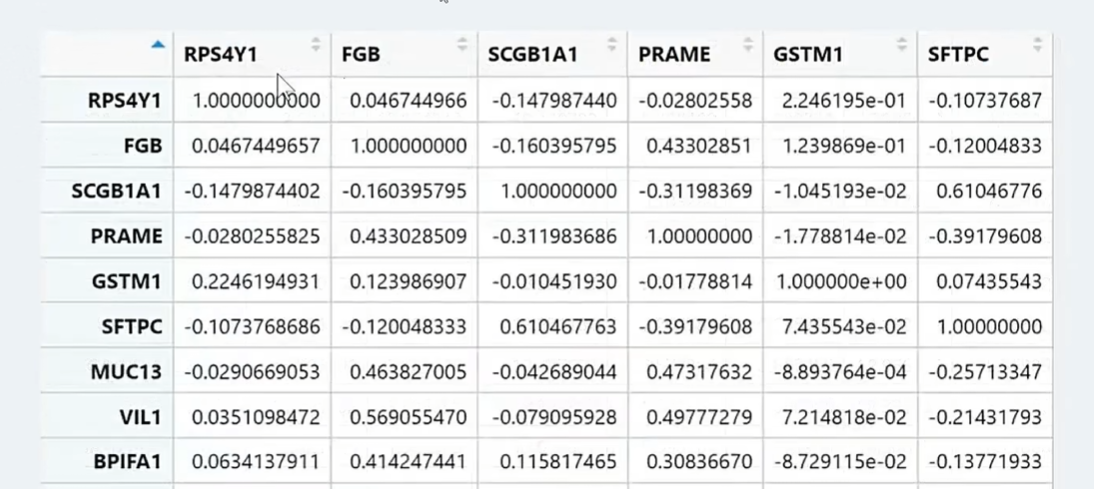
其中相关性表达矩阵表格中,例如RPS4Y1基因和自身的相关性为1,大于零为正相关,而小于零为负相关,构建完成相关性表达矩阵后就可以利用WGCNA去构建相关性表达网络
2.2相关性网络(需要构建什么样的网络?)
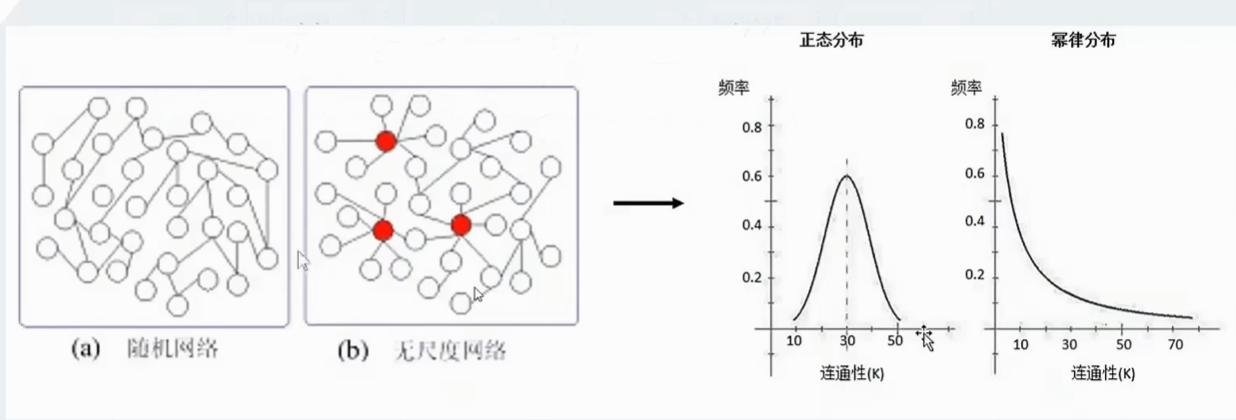
在网络中,节点代表基因,线代表相关性。如上图两种网络当中,我们要构建的的是无尺度网络,该网络服从幂律分布,即少数的节点的连接数较高,大部分节点连接数较低。而不是随机网络,该网路服从正态分布,即大多数节点的连接数较高,而少部分的连接数较低。原因是,在生物体内只有少数蛋白质或者基因参与大量的相互作用,形成信号枢纽的作用,无尺度网络中连接度较高的节点,即是我们研究的重点,其作为枢纽作用,研究价值更高。假如在随机网络中,是大多数蛋白质都参与大量的相互作用,这样的话不就成混乱体系了吗?
2.3选择合适的软阈值(Power,判断基因是否具有相关性阈值)
2.3.1软阈值和硬阈值的区别差异
为了衡量两个基因是否具有相似表达模式,一般需要设置阈值来进行筛选,高于阈值就认为是相似的,但是这种设置阈值的方法存在问题,假如设定阈值为0.8,那么基因间相关性为0.81和0.79很难说明是有显著差异的,即无法区分。这种设置阈值的方法称为硬阈值
为了解决上述难以区分显著差异的问题,因此WGCNA分析时,采用相关系数加权值,即对基因相关系数取N次幂,这样计算出的是基因相关性的权重大小,我们只需要选取合适的N次幂即可,这种设置阈值的方法称为软阈值。
其中硬阈值是简单二元化的“有无”关系,这种方法是一刀切的筛选,大于设定阈值就符合否则就不符合。这样分析出的结果更趋近于正态分布,构建的网络也更趋近于随机网络是不符合生物学规律的。而软阈值则是连续的“权重关系”,而非单纯的“有无”,这样分享出的结果更符合幂律分布,构建出的网络更符合无尺度网络,因为幂次运算会放大强相关的边,而弱化弱相关,如相关性0.9取幂次后更接近1,而0.5则更接近0,使富者更富,贫者更贫,这样就会符合幂律分布,而硬阈值一刀切的方式,使得分布更随机。
2.3.2软阈值的筛选
挑选出合适的软阈值
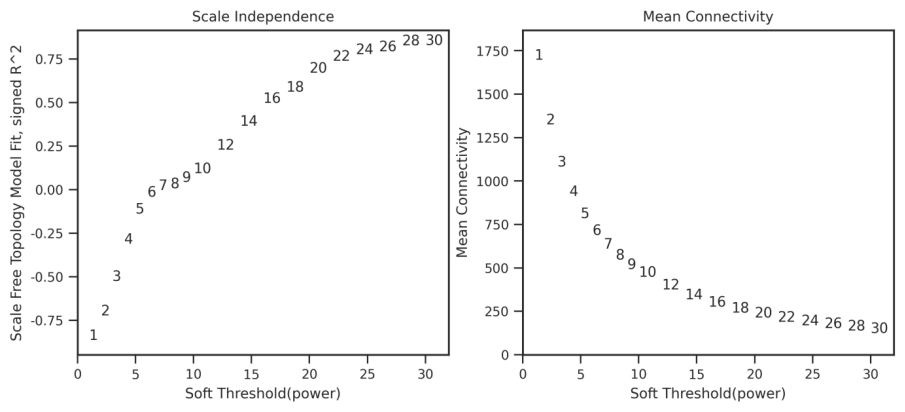
图一:无尺度拓扑拟合图,纵轴为拟合优度,横轴为Power值,拟合优度越趋近于1证明构成的网络越趋近于无尺度网络。
图二:平均连接度图,纵轴为平均连接度,横轴为Power值,在Power值增加的情况下,平均连接度会降低
判断软阈值的方法一般是根据这两个核心图来判断所需要的Power值,图一为无尺度拓扑拟合图,图二为平均连接度图。判断Power值的时候需要根据两个图一起判断,根据图一中的Power值与无尺度网络拟合度(R²)越高,但必须注意对应Power值对应在图二中的平均连接度不能太低。两者必须结合。



